Protista Ciliados: Uma Nova Fonte de Antibióticos
Cindy Prata Miranda & Marcus Vinicius Xavier Senra*
Centro de Ciências Naturais e Humanas, Universidade Federal do ABC, 09210-580, Santo André, São Paulo - Brasil.
*Autor para correspondência - Este endereço de email está sendo protegido de spambots. Você precisa do JavaScript ativado para vê-lo.; ORCID: 0000-0002-3866-8837
Resumo: O surgimento e disseminação de microrganismos multirresistentes aos antibióticos convencionais representa uma séria ameaça à saúde global, pois compromete a eficácia dos tratamentos médicos, aumenta a morbidade e mortalidade associadas a infecções e impõe desafios significativos aos sistemas de saúde. Nesse contexto, torna-se essencial buscar novas opções para o tratamento de infecções causadas por microrganismos resistentes. Uma alternativa promissora aos antibióticos convencionais são as defensinas estabilizadas por cisteínas do tipo αβ (defensinas CS-αβ), pequenos peptídeos antimicrobianos (AMPs) produzidos por plantas, animais e fungos, e utilizados como uma defesa natural contra ampla gama de patógenos, incluindo bactérias, fungos, vírus e protozoários. Desta forma, estamos aplicando diferentes estratégias de bioinformática e bancos de dados públicos de sequências biológicas para identificar e caracterizar novas defensinas, com potencial uso na terapêutica humana, veterinária e para fins industriais; e, ainda, estamos desenvolvendo uma nova e eficiente plataforma para produção destas pequenas moléculas com propriedades antibióticas, baseada em uma microalga, chamada Chlamydomonas reinhardtii, para viabilizar economicamente a chegada destas descobertas à população.
Palavras-chave: resistência antimicrobiana; patógenos; mineração genômica; Ciliophora; bioprospecção.
Abstract: The emergence and spread of microorganisms that are multi-resistant to conventional antibiotics represents a serious threat to global health, as it compromises the effectiveness of medical treatments, increases the morbidity and mortality associated with infections and poses significant challenges to health systems. In this context, it is essential to look for new options for treating infections caused by resistant microorganisms. A promising alternative to conventional antibiotics are cysteine-stabilized αβ defensins (CS-αβ defensins), small antimicrobial peptides (AMPs) produced by plants, animals, and fungi, which are used as a natural defense against a wide range of pathogens, including bacteria, fungi, viruses and protozoa. In this way, we are applying different bioinformatics strategies and public databases of biological sequences to identify and characterize new defensins, with potential use in human and veterinary therapeutics and for industrial purposes; and we are also developing a new and efficient platform for the production of these small molecules with antibiotic properties, based on a microalga called Chlamydomonas reinhardtii, in order to make it economically viable for these discoveries to reach the population.
Keywords: antimicrobial resistance; pathogens; genomic mining; Ciliophora; bioprospecting.
A descoberta e o uso terapêutico de agentes antimicrobianos representa um dos maiores marcos da ciência médica, contribuindo para salvar inúmeras vidas todos os anos1. Infelizmente, demorou apenas 7 anos, desde o início da comercialização do primeiro antibiótico, a penicilina, em 19412, até o aparecimento da primeira linhagem de Staphylococcus aureus resistente, um caso registrado em pacientes hospitalizados em Londres em 19483. Hoje, já há relatos de inúmeros casos preocupantes de microorganismos resistentes e multirresistentes aos mais diversos antibióticos comercialmente disponíveis, como Mycobacterium tuberculosis, Streptococcus pyogenes, Staphylococcus aureus, Enterobacter cloacae, Pseudomonas aeruginosa, Candida albicans e Aspergillus fumigatus, resultantes de processos de seleção natural, mas exacerbados pelo uso indevido e excessivo de antibióticos2. Portanto, o desenvolvimento de novos antimicrobianos é extremamente necessário para combater essa grande preocupação na saúde pública.
Nesta atual era Pós-Genômica em que vivemos, resultado da popularização e barateamento nos custos associados ao sequenciamento em larga escala de DNA, podemos perceber um aumento exponencial no número de genomas, tanto de organismos modelo, como de organismos não-modelo, sendo publicados e depositados em bancos dados de sequências. Tal dinâmica de publicação vem se tornando, portanto, uma rica fonte de informação biológica, de valor incalculável para auxiliar na descoberta de novas moléculas bioativas, incluindo novos antibióticos.
Dentre os organismos eucarióticos ainda pouco estudados, mas com dezenas de genomas disponíveis nestes bancos de dados, podemos citar diversas espécies que compõem o filo Ciliophora (Alveolata), um grupo protistas unicelulares e microscópicos com uma enorme diversidade (aproximadamente 8000 espécies descritas) e origem ancestral (1.1 Bilhão de anos), capaz de colonizar quase que qualquer ecossistema marinho e terrestre, onde desempenham importante função na alça microbiana, atuando como os principais predadores de bactérias e outros microorganismos [4]. Interessantemente, mesmo com esta íntima e complexa interação com outros microrganismos, não há relatos de patógenos letais em ciliados, sugerindo um eficiente mecanismo de controle de infecções, ainda muito pouco conhecido e explorado pela ciência.
Desta forma, o objetivo deste estudo realizado na Universidade Federal do ABC (UFABC) foi varrer os genomas de ciliados disponíveis, em busca de novas moléculas com propriedades antibióticas e iniciar o desenvolvimento de uma metodologia eficiente e comercialmente viável, utilizando uma microalga, chamada Chlamydomonas reinhardtii como uma plataforma de expressão, para produção em larga escala destas moléculas, que poderão vir a ser aplicadas no combate a estas muitas bactérias e fungos resistentes e multirresistentes aos mais diversos antibióticos convencionais.
Iniciamos este estudo recuperando os dados genômicos a partir de um banco internacional de sequências, chamado Genbank (https://www.ncbi.nlm.nih.gov/genbank/). Após o tratamento apropriado para garantir a qualidade dos dados, reconstruímos os genomas de 162 espécies de protistas ciliados e iniciamos uma varredura por novas moléculas com características antimicrobianas, utilizando, para isso, uma variedade de ferramentas de bioinformática, que incluíram: buscas por moléculas homólogas, ou seja, procurávamos por moléculas parecidas a outras já bem caracterizadas, disponíveis nos banco de dados de sequências. Uma descrição detalhada do procedimento e ferramentas de bioinformática aplicadas podem ser encontradas em um artigo recentemente publicado pelo grupo responsável por esta pesquisa [5]. Além disso, foi utilizado recursos de inteligência artificial (IA) para a identificação de novas moléculas. Para isso, treinamos um modelo de “deep learning” com um grande banco de dados composto por 1024 proteínas que sabidamente apresentam atividade antimicrobiana e 1024 proteínas que não apresentam tal atividade. De acordo com nossos testes cegos, o modelo possui uma boa capacidade preditiva (acurácia de 91,7%, uma sensibilidade de 93,9% e uma especificidade de 98,7%), o que nos deixa bastante confiantes quanto a sua eficiência em identificar bons candidatos codificados nos genomas destes organismos.
Ao aplicar esta metodologia, fomos capazes de identificar um total de 101 peptídeos, potencialmente com características antibióticas. Destes, 96 são pequenos peptídeos catiônicos (10-15 resíduos de aminoácidos) identificados por métodos de IA, que apresentam 99% de probabilidade de apresentarem atividade antimicrobiana. Além de 5 defensinas do tipo αβ estabilizadas por cisteínas, ou simplesmente defensinas CS-αβ [5]. Esses peptídeos antimicrobianos (AMPs) são geralmente pequenos (34-54 resíduos de aminoácidos), anfipáticos (contém regiões hidrofóbicas e hidrofílicas definidas na mesma molécula), principalmente catiônicos, ricos no aminoácido cisteína, e que adotam uma estrutura tridimensional canônica, consistindo em uma hélice α e duas ou três folhas β antiparalelas estabilizadas por três pontes de dissulfeto (ligação covalente entre dois resíduos de cisteínas) [6].
As defensinas, de um modo geral, podem ser encontradas em uma diversidade de fungos, plantas e em alguns invertebrados, onde exercem suas funções de defesa contra patógenos por meio de diferentes mecanismos de ação, incluindo indução à ruptura e/ou formação de poros em suas membranas, bloqueio de canais de íons ou interferência direta sobre diferentes vias intracelulares; sendo altamente eficazes contra um amplo espectro de patógenos, podendo apresentar atividade antifúngica, inseticida, bactericida, e também antiprotozoários [6].
Estes AMPs são ainda interessantes pois costumam apresentar baixos efeitos citotóxicos em células humanas, são termicamente e proteoliticamente estáveis, passíveis de engenharia racional e raramente induzem resistência adquirida em comparação com os antibióticos convencionais [6]. Além disso, muitas defensinas CS-αβ conhecidas, como a lucifensina [7], isolada de moscas varejeiras ( Lucilia sericata); e a scedosporisina [8], isolada de fungos (Scedosporidium apiospermum), são altamente eficazes contra patógenos humanos, como Staphylococcus aureus resistente à meticilina e Enterococcus spp. resistente à vancomicina, respectivamente, destacando que essas moléculas têm grande potencial para servir como alternativas aos antibióticos convencionais na terapêutica humana.
Na figura 1, apresentamos representações das estruturas tridimensionais de duas destas defensinas (LsAMP1 e LsAMP2), que caracterizamos em estudo recente [5], onde podemos observar a característica conformação CS-αβ destas moléculas. Ambas defensinas foram identificadas no genoma de um mesmo ciliado de de água doce, identificado como Laurentiella sp. e são muito parecidas (identidade de sequência > 65%) com defensinas antifúngicas já caracterizadas de alguns insetos lepidópteros (grupo que inclui as borboletas e mariposas), chamadas de heliomicina9 e ARD110 (Tabela 1). Dada esta semelhança estrutural, e por também apresentarem um mesmo domínio conservado, chamado de núcleo gama, responsável pela atividade antifúngica nesta classe de AMPs, acreditamos que estas duas defensinas de ciliados também tenham propriedades contra fungos. De fato, simulações adicionais feitas em supercomputadores sugerem que elas, muito provavelmente, utilizam um constituinte da membrana plasmática, chamado fosfatidilinositol 4,5-bifosfato (PIP2), como mecanismo de ancoragem e inserção na membrana plasmática de suas células-alvo [5].
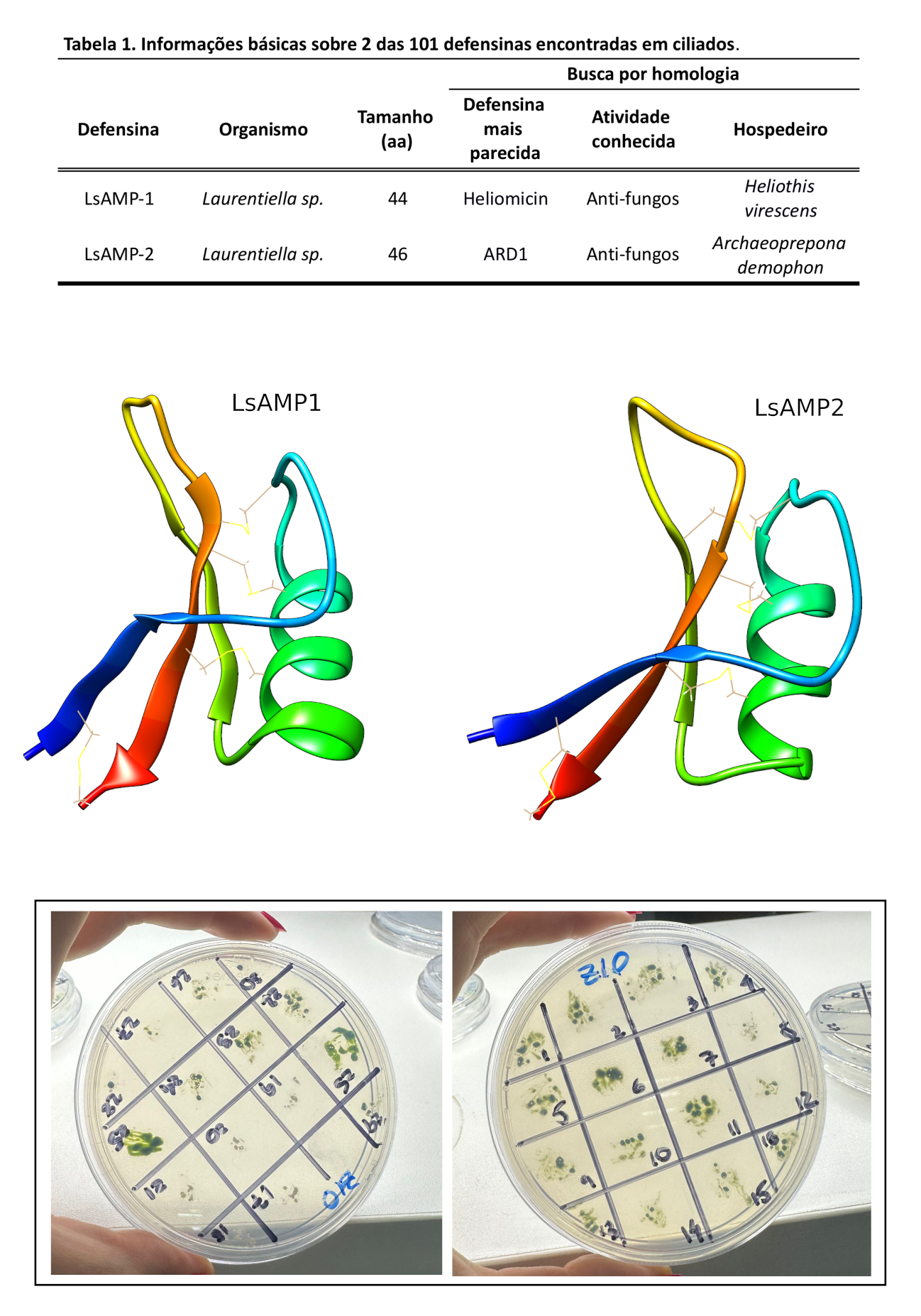
Figura 1. Dois peptídeos antimicrobianos (AMPs) (LsAMP1 e LsAMP2) identificados a partir de genomas de um ciliado, Laurentiella sp.; e linhagens de Chlamydomonas reinhardtii produtoras de um destes AMP (LsAMP1).
Contudo, eu ou qualquer um de nós podemos acreditar em qualquer coisa, só isso não é suficiente quando estamos falando de Ciência feita de um jeito sério. Nós precisamos ser capazes de determinar experimentalmente quem são os alvos e o mecanismo de ação destas defensinas, se quisermos aplicá-las à terapêutica humana, por exemplo. Desta forma, decidimos que o próximo passo em nosso estudo seria ensinar uma microalga, chamada Chlamydomonas reinhardtii, a produzir estas moléculas com propriedades antibióticas. Neste momento, você pode estar se perguntando: por que C. reinhardtii? Eu te explico.
A escolha desta microalga se deu por oferecer várias vantagens em relação a outros sistemas comumente utilizados em biotecnologia para produção de proteínas recombinantes. Como assim? Com frequência, estratégias de super-expressão em sistemas heterólogos (produzir uma proteína de um organismo em um outro organismo) são aplicadas utilizando bactérias, como Escherichia coli, ou fungos, como Pichia pastoris, por conta de suas altas taxas de duplicação, baixo custo de cultivo e diversidade de ferramentas genéticas já muito bem estabelecidas. Contudo, uma vez que estes organismos são, em geral, os principais alvos dos AMPs, existe uma clara necessidade por novos sistemas para a eficiente super-expressão de AMPs, que também proporcionem simultâneamente alto rendimento e baixo custo de produção, viabilizando, assim, sua exploração comercial.
Por isso, optamos pela Chlamydomonas reinhardtii, uma microalga que não é um alvo primário das defensinas e que representa um sistema já bem estabelecido e utilizado na produção em larga escala de diferentes tipos de moléculas, inclusive alguns fármacos. Além disso, estas microalgas apresentam alta taxa de crescimento, baixo custo de manutenção in vitro e contam com uma ampla diversidade de métodos de modificação genética e de expressão heteróloga de proteínas11.
Desta forma, utilizando técnicas de Biologia Molecular e de Biologia Sintética, estabelecemos linhagens de C. reinhardtii capazes de sintetizar uma variedade destas pequenas moléculas de interesse e, em breve, poderemos purificar e realizar testes in vitro para determinação de suas eficácias, espectro e mecanismo de ação, para que, aí sim, tenhamos dados suficientes de avaliação do potencial uso destas moléculas na terapêutica humana e de outros animais.
Para finalizar, nosso trabalho enfatiza a importância de bancos de dados de sequências públicas e métodos baseados em computação para estudos de bioprospecção e descoberta de novos medicamentos, oferecendo uma estrutura rápida e de baixo custo para a identificação de novos antimicrobianos; bem como a necessidade de se desenvolver novas plataformas de expressão de proteínas recombinantes, a fim de atender as particularidades de cada tipo de moléculas de interesse biotecnológico. Além disso, esses 101 novos AMPs que identificamos a partir dos genomas de diferentes espécies de ciliados de vida livre destacam que esses organismos, de certa forma negligenciados pela Ciência, podem se tornar uma importante fonte de novas de moléculas bioativas com uso potencial na terapêutica humana e veterinária, mas também com aplicações em plantas de interesse agronômico e para atender a crescente indústria de alimento e cosméticos.
Agradecimentos
Este estudo foi financiado por um Auxílio Regular FAPESP (2022/00538-0) e inclui dados obtidos durante a vigência da bolsa de Iniciação Científica (PDPD/2022), concedida pela UFABC à Cindy Prata. Gostaríamos de agradecer ainda à Central Computacional Multiusuário da Pró-Reitoria de Pesquisa da UFABC, pela disponibilização de horas de uso no supercomputador, Titânio, que foi fundamental para realização de todas as nossas análises e simulações in silico. Agradecemos, ainda, à professora Lívia Seno (UFABC) pela amizade e pelo apoio fundamental para o início deste estudo.
Referências bibliográficas
1. Davies, J. & Davies, D. Origins and Evolution of Antibiotic Resistance. Microbiol Mol Biol Rev. 74:417–33 (2010).
2. Levy, S.B., Bonnie, M. Antibacterial resistance worldwide: causes, challenges and responses. Nat Med. 10:122–9 (2004).
3. Barber, M., Rozwadowska-Dowzenko, M. Infection by penicillin-resistant Staphylococci. Lancet. 252:641–4 (1948).
4. Lynn, D. The Ciliated Protozoa. 3rd edition. New York, NY: Pergamon Press (2008).
5. Senra, M.V.X. In silico characterization of cysteine-stabilized αβ defensins from neglected unicellular microeukaryotes. BMC Microbiol. Mar 25;23(1):82 (2023).
6. Dias, R.D.O., Franco, O.L. Cysteine-stabilized αβ defensins: from a common fold to antibacterial activity. Peptides. 72:64–72 (2015).
7. Valachova, I., Prochazka, E., Bohova, J., Novak, P., Takac, P., Majtan, J. Antibacterial properties of lucifensin in Lucilia sericata maggots after septic injury. Asian Pac J Trop Biomed. 4:358–61 (2014). 8. Wu, J., Liu, S., Wang, H. Invasive fungi-derived defensins kill drug-resistant bacterial pathogens. Peptides. 99:82–91 (2018).
9. Lamberty, M., Caille, A., Landon, C., Tassin-Moindrot, S., Hetru, C., Bulet, P., et al. Solution structures of the antifungal heliomicin and a selected variant with both antibacterial and antifungal activities. Biochemistry. 40:11995–2003 ( 2001).
10. Landon, C. Lead optimization of antifungal peptides with 3D NMR structures analysis. Protein Sci. 13:703–13 (2004).
11. Arias, C.A.D., Oliveira, C.F.M.D., Molino, J.V.D., Ferreira-Camargo, L.S., Matsudo, M.C., Carvalho, J.C.M.D. Production of Recombinant Biopharmaceuticals in Chlamydomonas reinhardtii. International Journal of Plant Biology. 14(1):39-52 (2023).

Redes Sociais